本稿は TeX & LaTeX Advent Calendar 2017 の2日目の記事です.1日目は ZR さんでした.3日目は golden_lucky さんです.
Twitter 上や当ブログ上において,私(@wtsnjp)は完全に「TeX の人」として認識されているのではないかと危惧しているのですが,実は大学では生物情報科学(バイオインフォマティクス)という分野を専攻しています1.
もう1年以上も前のことですが,大学の講義で生物情報科学の分野では非常に有名な Gotoh アルゴリズム(Smith-Waterman-Gotoh アルゴリズム)を実装する演習課題がありました.この課題では使用言語は特に指定されていなかったため,私は誰もが近寄りたがらない素晴らしい TeX 言語を用いて実装を行いました2.
このとき作成した TeX 言語コードは,提出後に LaTeX パッケージとして実用に耐え得る形に整理して,Package Gotoh という名前で公開しています.CTAN にもアップロード済みなので新しい TeX Live には標準で添付されています.
- GitHub: https://github.com/wtsnjp/gotoh
- CTAN: https://ctan.org/pkg/gotoh
この件については,既に国際会議での発表や論文の投稿を行ってありますが(詳細後述),日本語の情報はこれまでほとんど提供してこなかったので,この機会に簡単にご紹介してみようと思います.
配列アライメント
生物学,特に生物情報科学の分野においては複数の DNA 配列間の「類似性」や「距離」を考えることが非常に重要です.そのため,生物情報科学では配列アライメントと呼ばれる問題がしばしば取り扱われます.
配列アライメントは2以上の任意の数の配列についての問題ですが,2つの配列のアライメントを特にペアワイズアライメントといいます.これは,簡単に言うと「2つの配列(文字列)をなるべく似ている部分が重なるように並べる操作」です.このことを厳密に定式化するのは煩雑なので,ここでは具体例を挙げて説明します.
配列 “GACTA” と “GAGA” のペアワイズアライメントを考えると,両者は長さが違いますが,最初の2文字(GA)と最後の1文字(A)は一致しています.そこで,これらの共通部分が重なるようにするには,次のように並べることが考えられます.
| 1 | 2 | 3 | 4 | 5 |
|---|---|---|---|---|
| G | A | C | T | A |
| G | A | . | G | A |
ここで1, 2, 5列目は両者の文字が一致しています.この状態をマッチといいます.3列目は下の配列に . が差し込まれています.生物学的には,この位置で塩基の欠失(または上の配列への塩基の挿入)が起こったと解釈されるわけですが,これをギャップと呼びます.4列目は上と下の文字が異なっており,これはミスマッチと呼ばれます.もちろん,生物学的にはこの位置で塩基の置換が起こったと解釈するのが普通でしょう.
ペアワイズアライメントとは,先ほどとは別の言い方をすると「2つの配列をギャップやミスマッチがなるべく少なくなるように並べる操作」ということになります.
Gotoh パッケージ
それぞれ $m, n$ の長さをもつ2つの配列について,そのペアワイズアライメントを $O(mn)$ 時間で計算するアルゴリズムが Gotoh アルゴリズムです.そして,これを TeX 言語で実装したものが拙作の Gotoh パッケージです.
このパッケージのインターフェースは極めてシンプルで,アルゴリズムを実行する \Gotoh 命令と各種パラメタ設定を行う \GotohConfig コマンドの2つしかありません.
\Gotoh 命令の基本的な書式は次の通りです.
\Gotoh{<sequence A>}{<sequence B>}
\Gotoh 命令によって実行されたアライメントの結果は \GotohResultA と \GotohResultB に返ります.したがって
\documentclass{article}
\usepackage{gotoh}
\begin{document}
\begin{flushleft}
\Gotoh{ATCGGCGCACGGGGGA}{TTCCGCCCACA}
\texttt{\GotohResultA} \\
\texttt{\GotohResultB}
\end{flushleft}
\end{document}
という LaTeX ソースは次のような印刷結果となります.
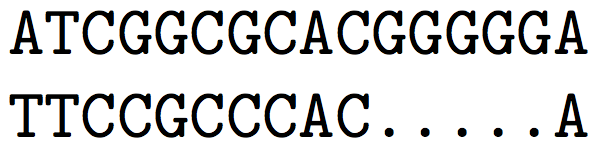
なお,結果を返す制御綴は \GotohConfig 命令によってお好きなものに変更可能です.
\GotohConfig{
result A = \ShiroYukidaruma,
result B = \KuroYukidaruma}
\GotohConfig コマンドでは他にも配列の大文字化を行うか否かや,アライメント計算時に用いられるパラメタの値などを設定することも可能です.
各命令の詳しい仕様については付属ドキュメントを参照してください3.
TeXshade との連携
TeX & LaTeX Advent Calendar 2014 の17日目,私が初めてこのアドベントカレンダーに参加した際の記事は次のようなものでした.
この記事の中で TeXshade という,外部の専用計算ソフトウェアによって得られた配列アライメントの「結果」を見やすく綺麗に印刷するためのパッケージを紹介しています.したがって当然,Gotoh パッケージと連携することも可能です.
現状 TeXshade パッケージには LaTeX ソース中から直接アライメント結果を入力する方法が存在しないため,一旦 *.fasta という外部ファイルに結果を出力させてから TeXshade のインターフェースを用いて再びこれを読み込んでやる必要があります.この操作をルーチン化したのが次のマクロです.
% output file
\newwrite\FASTAfile
\def\writeFASTA#1{%
\immediate\write\FASTAfile{#1}}
% print alignment
\newcommand{\PrintAlignment}[3][\relax]{%
\Gotoh{#2}{#3}%
\immediate\openout\FASTAfile=\jobname.fasta
\writeFASTA{> Seq 1^^J\GotohResultA}%
\writeFASTA{> Seq 2^^J\GotohResultB}%
\immediate\closeout\FASTAfile
\texshade{\jobname.fasta}#1\endtexshade}
ここで定義されている \PrintAlignment 命令は次のような書式を持っています.
\PrintAlignment[<TeXshade commands>]{<sequence A>}{<sequence B>}
1つ目のオプション引数には TeXshade の表示結果を設定するための命令4を列挙することができます.残りの引数は \Gotoh 命令と同様です.
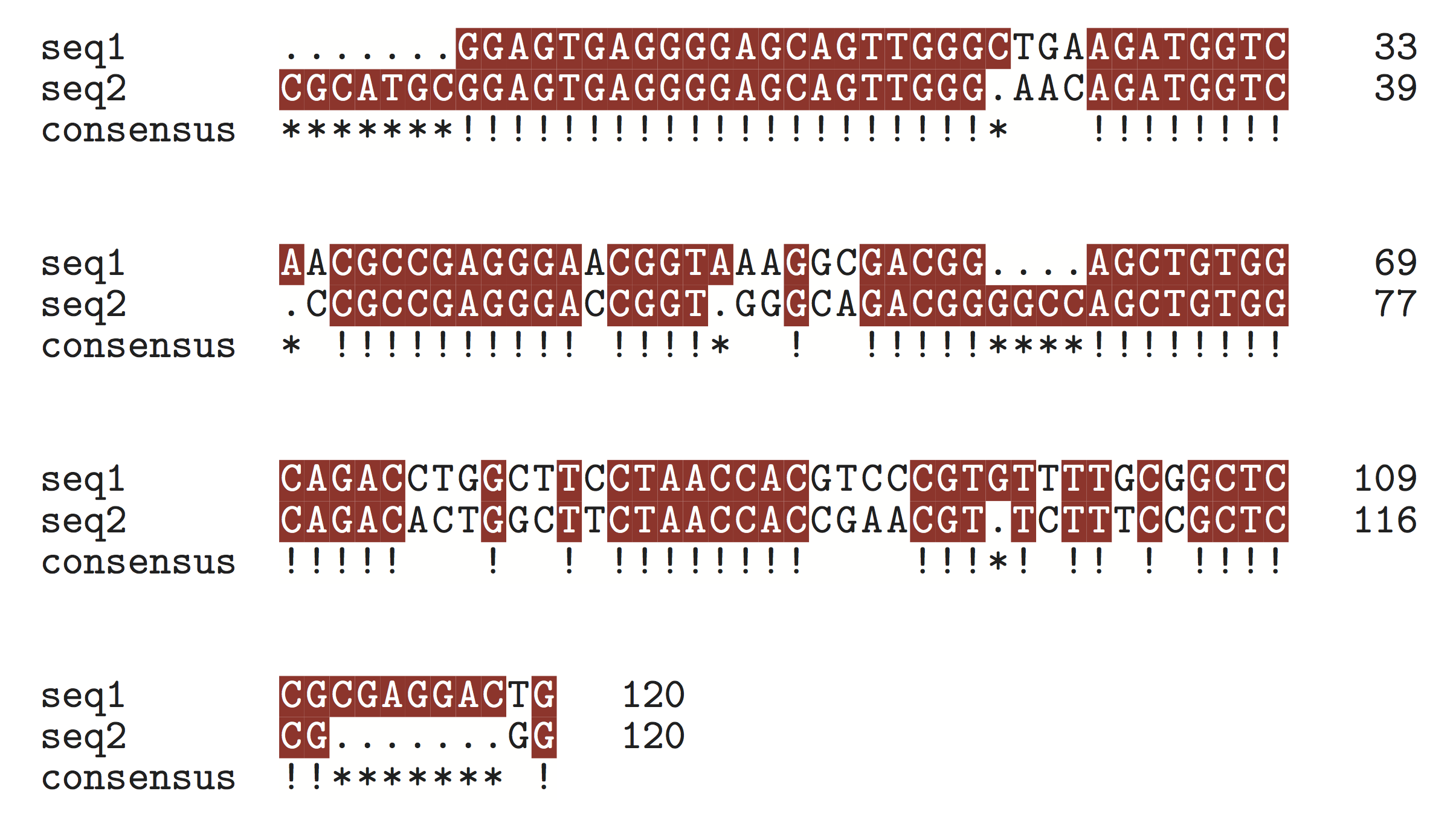
これによって,配列アライメントの計算から結果の出版品質での印字まで,すべてを TeX だけで完結することが可能になりました.以下はそのサンプル LaTeX 文書です.
\documentclass{article}
\usepackage{gotoh}
\usepackage{texshade}
%% output file
\newwrite\FASTAfile
\def\writeFASTA#1{%
\immediate\write\FASTAfile{#1}}
%% print alignment
\newcommand{\PrintAlignment}[3][\relax]{%
\Gotoh{#2}{#3}%
\immediate\openout\FASTAfile=\jobname.fasta
\writeFASTA{> Seq 1^^J\GotohResultA}%
\writeFASTA{> Seq 2^^J\GotohResultB}%
\immediate\closeout\FASTAfile
\texshade{\jobname.fasta}#1\endtexshade}
\begin{document}
\PrintAlignment[{\shadingcolors{reds}}]
{GGAGTGAGGGGAGCAGTTGGGCTGAAGATG%
GTCAACGCCGAGGGAACGGTAAAGGCGACG%
GAGCTGTGGCAGACCTGGCTTCCTAACCAC%
GTCCCGTGTTTTGCGGCTCCGCGAGGACTG}
{CGCATGCGGAGTGAGGGGAGCAGTTGGGAA%
CAGATGGTCCCGCCGAGGGACCGGTGGGCA%
GACGGGGCCAGCTGTGGCAGACACTGGCTT%
CTAACCACCGAACGTTCTTTCCGCTCCGGG}
\end{document}
TUG@BachoTeX 2017 での発表と TUGboat への投稿
今年の4月末から5月の頭にかけて,ポーランドにて TUG@BachoTeX 2017 という TeX に関する国際的なカンファレンスが開催されました.私は初の単独海外渡航でこの会議に参加して,この Gotoh パッケージを開発したことについて口頭発表を行いました(そして幸運にも W. J. Martin Prize という賞をいただきました).
また,同発表の内容については TUG (TeX Users Group) の発行する TUGboat という TeX 専門ジャーナルに講演と同じ “Implementing bioinformatics algorithms in TeX ― the Gotoh package, a case study” というタイトルで論文の投稿もしています(TUGboat 38:2, 2017).
本稿の駆け足な説明では物足りないという読者の皆さんは,ぜひこれらにも目を通していただけると嬉しいです.
-
来年からは現所属大学の外に出て,自然言語処理を専門として研究を行っていく予定です.ちなみに,現在取り組んでいる卒業研究も実際には生物情報科学や生物学とはほとんど関係のない自然言語処理関連の仕事です. ↩︎
-
正確には「難解プログラミング言語」は不可との指示がありましたが,以前にも言及したように TeX 言語は難解プログラミング言語ではありません. ↩︎
-
付属ドキュメントではアルゴリズムや実装についても詳しく解説してあります(ただし英語のみです).新しい TeX Live をご利用の場合は
texdoc gotohで閲覧可能です. ↩︎ -
TeXshade に関しては,先述した拙作の記事がほぼ唯一の日本語文献です.より詳しい情報については
texdoc texshadeによって付属ドキュメントを参照してください. ↩︎